
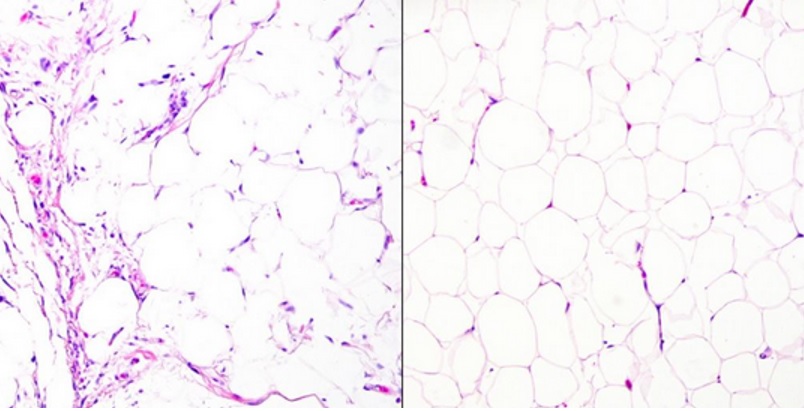
C’est un test plus rapide, moins cher et plus précis permettant de détecter les liposarcomes, des tumeurs difficiles à diagnostiquer, qui vient d’être développé par une équipe de pathologistes de l’University of British Columbia (Vancouver). Documenté dans le Journal of Molecular Diagnostics, le test permet de différencier de manière précise un liposarcome (à gauche sur visuel) vs un lipome bénin (à droite) grâce au recours aux dernières technologies de séquençage ARN et d'apprentissage automatique.
« Les liposarcomes sont un type de cancer malin difficile à diagnostiquer car, même au microscope, il est difficile de différencier les liposarcomes de tumeurs bénignes ou d'autres types de cancer qui nécessitent des traitements différents », explique l’auteur principal, le Dr Torsten Owen Nielsen, du Centre de pathologie génétique de l’Université de la Colombie-Britannique. En effet, de nombreux liposarcomes ressemblent à leurs homologues bénins et relativement communs, les lipomes. Dans certains cas, l’incertitude diagnostique peut entraîner un stress sévère pour les patients et un diagnostic erroné peut avoir de nombreuses conséquences, dont le retard du traitement ou a contrario une chirurgie inutile.
Ce nouveau test basé sur un ensemble (ou panel) de gènes distingue rapidement et de manière fiable les lipomes bénins des liposarcomes et peut être réalisé en laboratoire à un coût inférieur à celui des tests actuels standards.
La technologie NanoString pour un diagnostic plus rapide et plus rentable des liposarcomes
Utiliser les 20 gènes les plus courants surexprimés dans les liposarcomes : parmi les tests diagnostiques actuellement recommandés pour les liposarcomes, l'immunohistochimie (IHC) est parfois inexacte et difficile à interpréter, et l'hybridation in situ par fluorescence (FISH) est relativement coûteuse.
Ici, les chercheurs canadiens regardent si une technologie nommée « NanoString », adaptée à l’analyse de l'ARN de mauvaise qualité, peut permettre un diagnostic plus rapide et plus rentable des liposarcomes via l'expression génique.
Les chercheurs ont utilisé les données du Cancer Genome Atlas, une base de données génétiques provenant de plus de 20.000 échantillons de cancer, pour identifier les 20 gènes les plus courants surexprimés dans les liposarcomes. Des sondes pour ces gènes ont été conçues par la bioinformatique NanoString et appliquées à un ensemble « d'échantillonsd’apprentissage » qui comprenait des échantillons de lipomes et des échantillons de liposarcomes. L'analyse des résultats permet de distinguer clairement les cas de lipome des cas de liposarcome.
Un modèle d'apprentissage automatique a été développé pour déterminer la probabilité qu'un échantillon donné soit positif pour le liposarcome et a ensuite été appliqué à 45 cas rétrospectifs pour déterminer les limites des prédictions positives et négatives. Le test a ensuite été testé en environnement clinique réel : un technicien a été invité à identifier chaque échantillon comme « liposarcome » ou « non liposarcome » à partir du test NanoString. Les mêmes échantillons ont été examinés par des pathologistes spécialisés en utilisant les tests standard.
- Le test NanoString montre une précision de 93% et est en accord avec les tests standard dans 97,8% des cas. Les résultats du test NanoString sont de plus disponibles en 36 heures, alors que les autres tests demandent un délai d’1 à 2 semaines. Enfin, le test NanoString apparaît beaucoup moins coûteux que les tests sandards.
Les progrès de la biotechnologie se sont révélés très prometteurs dans d'autres cancers tels que le cancer du sein. De plus, il n'y a pas de brevet sur ce test, ajoutent les chercheurs : « N'importe quel médecin peut appliquer la méthode que nous décrivons ».
L’équipe travaille actuellement à élargir ce test à d’autres types de sarcome et à d'autres types de cancer.
Source: The Journal of Molecular Diagnostics December 2020 DOI: 10.1016/j.jmoldx.2020.11.011 A Rapid and Cost-Effective Gene Expression Assay for the Diagnosis of Well-Differentiated and Dedifferentiated Liposarcomas
Plus sur Cancer Blog et Diagnostic Blog
